Zakład Zasobów Rybackich prowadzi szeroki zakres badań, mających na celu racjonalne gospodarowanie zasobami ryb. Naukowa działalność zakładu skupia się przede wszystkim na badaniu dynamiki populacji ryb użytkowych, analizujemy wpływ czynników środowiskowych na ryby oraz monitorujemy ich choroby. W ramach zakładu NB funkcjonuje również laboratorium genetyczne, w którym prowadzone są badania z zakresu genetyki populacji ryb użytkowych. Wspomagają one proces szacowania zasobów poprzez identyfikowanie rozmieszczenia i zakresu występowania poszczególnych populacji ryb. Dziś chcemy Wam przybliżyć specyfikę pracy wykonywanej w laboratorium – w pomieszczeniu 819. Prześledzimy zatem drogę, jaką przechodzi materiał genetyczny (DNA/RNA) począwszy od komórek żywego organizmu aż do momentu uzyskania surowych danych – czyli ustalonego genotypu czy odczytanej sekwencji DNA/RNA. Przedstawimy także zakres realizowanych badań genetycznych, omawiając pokrótce ich praktyczne zastosowanie.
Izolacja Materiału Genetycznego
Na wstępnie należy podkreślić, że praca w laboratorium genetycznym wymaga wyjątkowej precyzji i czystości. DNA jest niewidoczne gołym okiem, a ilości materiału (roztworu) jakimi operujemy to najczęściej tysięczne lub milionowe części litra (video 1).
Proces izolacji materiału genetycznego to podróż, która zaczyna się od fragmentów płetw ryb, a czasem też od otolitów (twardych struktur w uchu wewnętrznym ryb) lub łusek. Dzięki możliwości zastosowania takiego materiału, możemy analizować nie tylko świeże próbki, ale również materiały archiwalne, sięgając do zasobów na przykład z lat 60’ ubiegłego stulecia.
Pierwszym krokiem jest homogenizacja próbek w specjalnym buforze. Polega ona na mechanicznej lub chemicznej dezintegracji tkanki, co skutecznie uwalnia DNA z komórek. Dodany na tym etapie enzym – proteinaza K rozkłada białka błon komórkowych (Fot. 1), termoblok (Fot. 2) zapewnia optymalną temperaturę reakcji enzymatycznej, a worteks (video 2) energicznie wytrząsając mieszaninę mechanicznie rozbija błony komórkowe.
 |
 |
||
| Fot. 1. 1 mililitr Proteinazy K służy do zhomogenizowania około 50 prób. | Fot. 2. Termoblok ogrzewający w próbki w trakcie homogenizacji |
W wyniku tego procesu DNA zostaje uwolnione do buforu, tworząc jednolitą mieszaninę z innymi związkami organicznymi i nieorganicznymi, z której można dalej DNA separować. Do tego procesu używamy kolumienek krzemionkowych (Fot. 3,4). Działają one jak precyzyjne filtry, selektywnie wiążąc jedynie kwasy nukleinowe i oddzielając je od innych składników komórkowych. Po kilku etapach odpłukiwania innych związków następuje proces elucji DNA. Eluowanie polega na przepuszczaniu przez kolumnę specjalnego buforu, który wypłukuje z niej związane DNA. Dzięki temu uzyskujemy czysty materiał genetyczny gotowy do dalszych analiz. Ze względu na minimalne ilości pozyskiwanego DNA spektrofotometr NanoDrop (Fot. 5) jest niezwykle przydatnym instrumentem.
 |
 |
 |
| Fot. 3. Mikrokolumienka ze złożem krzemionkowym (biała warstwa w elemencie po lewej) używana do izolacji materiału genetycznego. | Fot. 4. Nie trudno się domyślić, że jednym z najczęściej używanych narzędzi w pracach genetycznych jest mikropipeta automatyczna. | Fot. 5. Spektrofotometr NanoDrop One – pomiar stężenia i czystości DNA/RNA. |
Precyzyjnie ocenia stężenie i czystość DNA w 1 ul -kropli roztworu (Fot. 6). Wynik tej analizy jest kluczowy dla sukcesu kolejnych etapów badań, gdyż wysoka jakość i odpowiednia ilość DNA determinują możliwość przeprowadzania i wiarygodność dalszych procedur.
Łańcuchowa Reakcja Polimerazy (PCR)
Po izolacji materiału genetycznego dysponujemy zbyt małą ilością DNA, by przeprowadzić szczegółowe analizy. Aby uzyskać więcej DNA, wykorzystujemy technikę zwaną PCR. Pozwala ona na wielokrotne powielenie interesujących nas fragmentów DNA. Proces ten odbywa się w termocyklerze (Fot. 7), który zapewnia błyskawiczne zmiany i precyzyjną kontrolę temperatury, wymagane do przeprowadzenia reakcji PCR. Zachodzi ona w trzech etapach. Najpierw wysoka temperatura rozdziela spiralnie skręcone dwie nici DNA w procesie nazywanym denaturacją. Następnie, w niższej temperaturze, specyficzne startery – krótkie, wybrane przez nas sekwencje DNA, przyłączają się do wybranych miejsc na nici DNA – ten etap nazywa się anilingiem. W ostatniej fazie temperatura jest podwyższana, aby polimeraza DNA (enzym syntetyzujący nowe nici DNA) mogła wykonać swoje zadanie w procesie zwanym elongacją. Te cykle są powtarzane wielokrotnie, aby uzyskać miliony kopii DNA. Amplifikacja może obejmować zarówno geny, jak i inne niekodujące fragmenty DNA np. często używane w naszych analizach markery mikrosatelitarne (krótkie, powtarzające się sekwencje DNA). Mikrosatelity są szczególnymi markerami mającymi zastosowanie w analizie zróżnicowania genetycznego i struktury populacji, ponieważ charakteryzują się wysoką zmiennością. Pozwala to na odróżnienie pojedynczych organizmów czy populacji. Niektóre badania wymagają ustalenia na początku ilości kopiowanego DNA – do tego służy zakupiony w poprzednim roku aparat do ilościowej reakcji PCR czyli qPCR (Fot. 8).
 |
 |
 |
| Fot. 6. Do określenia ilości DNA wystarcza 1,5 mikrolitra płynu (kropla wielkości główki szpilki). | Fot. 7. Termocykler kontrolujący temperaturę w trakcie trwania reakcji PCR. | Fot. 8. Aparat do qPCR czyli ilościowej reakcji PCR |
Elektroforeza
Czasem kolejne kroki analizy DNA wymagają wizualizacji materiału genetycznego. Jednym ze sposobów jest rozdział powielonego DNA na żelu agarozowym podczas procesu zwanego elektroforezą. Żel działa jak sito molekularne, które rozdziela fragmenty DNA według ich wielkości. Pod wpływem prądu elektrycznego, mniejsze fragmenty DNA poruszają się szybciej od większych, migrując w żelu od bieguna ujemnego do dodatniego (Fot. 9). Po zakończeniu elektroforezy DNA jest obrazowane w świetle UV przy użyciu specjalistycznego aparatu do dokumentacji żeli firmy Bio-Rad (Fot. 10). Fluorescencyjnie zabarwione fragmenty DNA są wyraźnie widoczne, co umożliwia dokładne określenie ich pozycji. W celu ustalenia wielkości badanych fragmentów DNA na ścieżkę w żelu dodaje się marker wielkości, który w rozdziale elektroforetycznym przypomina charakterystyczną drabinkę. Jest to zestaw fragmentów DNA o znanych długościach (Fot. 11).
 |
 |
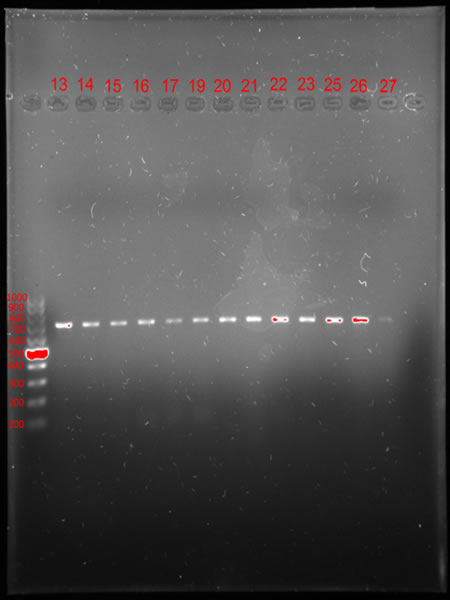 |
| Fot. 9. Aparat do elektroforezy w żelu agarozowym. | Fot. 10. Aparat do obrazowania wyników rozdziałów elektroforetycznych | Fot.11. Rozdział DNA podczas elektroforezy agarozowej – prążki markera wielkości (po lewej) umożliwiają określenie wielkości badanego nas fragmentu DNA w parach zasad (ścieżki 13-27). |
Dalsze Analizy
Po powieleniu fragmentów DNA, kolejnym krokiem jest ich genotypowanie lub sekwencjonowanie. Genotypowanie powielonego fragmentu DNA/RNA polega na przykład na precyzyjnym ustalaniu jego długości (w jednostkach zwanych parami zasad – Fot. 12).
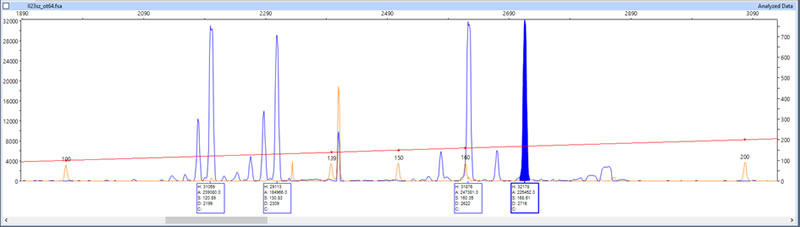 Fot. 12. Wynik genotypowania obrazujący długości czterech fragmentów DNA szczupaka.
Fot. 12. Wynik genotypowania obrazujący długości czterech fragmentów DNA szczupaka.
Sekwencjonowanie natomiast pozwala na odczytanie dokładnej sekwencji nukleotydowej w powielonych fragmentach. Często dopiero na tym etapie widzimy efekt naszej pracy, czyli czy udało się uzyskać odpowiednie dane genetyczne, czy też nie. Dopiero teraz dają o sobie znać potencjalne komplikacje, takie jak zanieczyszczenia próbek lub niedokładne przyłączenie się starterów podczas reakcji PCR. To na podstawie tych danych przeprowadzane są dalsze analizy i interpretacje.
Uzyskane dane pozwalają nam na rozwiązywanie szeregu problemów badawczych związanych z zarządzanie populacjami ryb i ochroną bioróżnorodności. Podstawowym naszym działaniem jest ustalanie struktury populacyjnej poszczególnych gatunków ryb, czyli dostarczamy informacji na temat geograficznego zasięgu i zróżnicowania genetycznego między populacjami danego gatunku. Określamy także poziom zmienności genetycznej w badanych populacjach. Wysoka różnorodność genetyczna jest często wskaźnikiem zdrowia populacji i miarą jej zdolności do przetrwania w obliczu zmian środowiskowych lub presji ze strony rybołówstwa. Informacje odnoszące się do poziomu zmienności genetycznej oraz wykonywane przez nas analizy potwierdzające przynależność do danej populacji są też niezbędne podczas prowadzenia programów zarybieniowych czy restytucyjnych – pozwalają na dobranie odpowiedniej puli genowej osobników używanych do tarła. Weryfikujemy efektywność prowadzonych zarybień (np. w prowadzonych od lat zarybieniach trocią polskich rzek), identyfikując ryby w rzekach pochodzące ze sztucznego tarła i te z naturalnego rozrodu. Prowadzimy też identyfikację gatunkową ryb i ich hybryd, w sytuacjach gdy metody morfometryczne nie są w stanie dać precyzyjnej odpowiedzi lub gdy mamy do czynienia z produktem spożywczym, dla którego skład gatunkowy chcemy potwierdzić. Identyfikacja gatunkowa może też dotyczyć pasożytów występujących w rybach, ale może być rozszerzona ogólnie na organizmy morskie. W ubiegłym roku zaczęliśmy włączać się w analizy środowiskowe, monitorując obecność oraz ilość złotych alg w wodach Zalewu Szczecińskiego. Z roku na rok się rozwijamy, starając się angażować w nowe problemy badawcze i współpracując z różnymi zakładami w obrębie MIR-PIB.
Remigiusz Szymański
Anna Wąs-Barcz


